
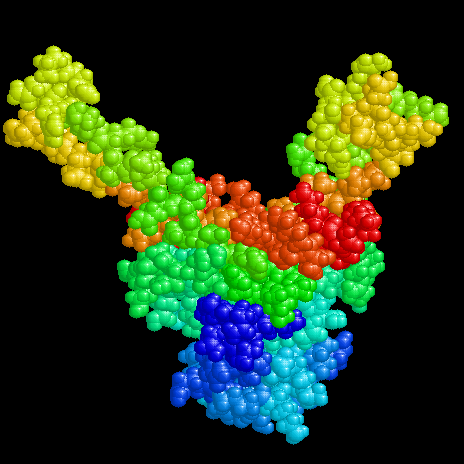
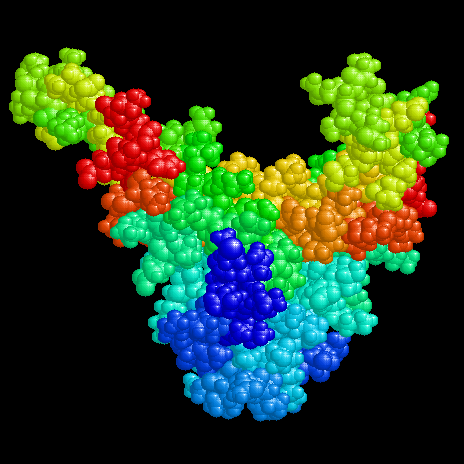
Die lineare Bilderbuchstruktur von DNA
ist weder für die Aufbewahrung in einer Zelle noch für die Funktion bei Transcription
und Replikation geeignet. In lebenden Zellen muß die DNA gefaltet werden, was in
Eukaryoten z.B. mittels Nucleosomen geschieht. Auch das ca 1mm lange Chromosom von
Escherichia coli muß kompaktiert werden, um in einem Raum von 1µm Durchmesser
unterzukommen. In Bakterien wurde eine Reihe von Histon-ähnlichen Proteinen gefunden,
die diese Aufgabe erfüllen sowie bei der Regulation von Transcription und Replikation
eingreifen. Von einigen dieser Proteine ist die molekulare Struktur aus
Röntgenbeugung von Kristallen oder NMR-Daten in Lösung bekannt.
Die hier vorgestellten Proteine sind ca. 20kDa groß und existieren als homologe
Heterodimere (IHF) oder Homodimere (HU, TF1). Sie binden unspezifisch an DNA (HU)
oder an definierte Konsensussequenzen (IHF, TF1). Die Strukturen sind durch kompakte
"Körper" gekennzeichnet, aus denen "Arme" herausragen, die mit der DNA wechselwirken.
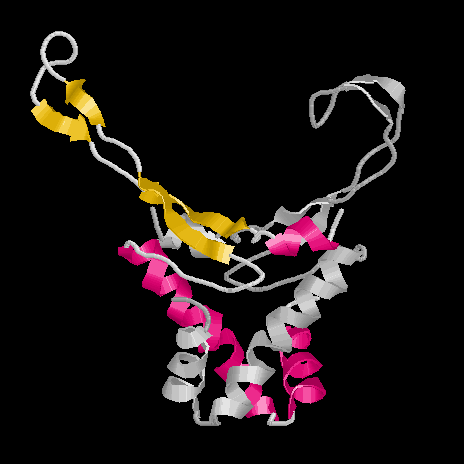 Dieses Bild von HU zeigt eine Untereinheit in grau, die andere gefärbt nach
Strukturmerkmalen: rot sind die alpha-Helices, die den "Körper" bilden, gelb die
beta-Stränge der "Arme". Die Arme enthalten jeweils zwei beta-Faltblätter mit einer
flexiblen Region dazwischen, dadurch wird ein Einfalten der Arme in die minor-groove
der DNA ermöglicht. Diese Struktur beruht auf Daten des gelösten Proteins ohne
gebundene DNA.
Dieses Bild von HU zeigt eine Untereinheit in grau, die andere gefärbt nach
Strukturmerkmalen: rot sind die alpha-Helices, die den "Körper" bilden, gelb die
beta-Stränge der "Arme". Die Arme enthalten jeweils zwei beta-Faltblätter mit einer
flexiblen Region dazwischen, dadurch wird ein Einfalten der Arme in die minor-groove
der DNA ermöglicht. Diese Struktur beruht auf Daten des gelösten Proteins ohne
gebundene DNA.
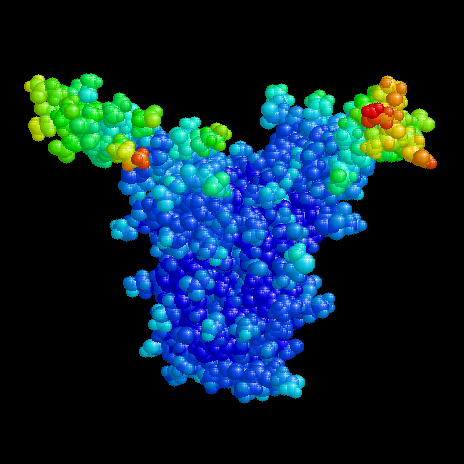 Dieses Bild von TF1 demonstriert die Beweglichkeit der Arme. Die Daten wurden
durch Röntgenbeugung eines Proteinkristalles gewonnen. Im Kristall hat jedes Atom
eine Unschärfe, die aus relativ unterschiedlichen Lagen in den individuellen
Molekülen resultiert. Diese Unschärfe ist in der Proteindatenbank als
"Temperaturfaktor" abgelegt. Im Bild sind "kalte" Atome (= geordnet, unbeweglich)
blau dargestellt; je "wärmer" eine Gruppe ist, desto "roter" ist sie abgebildet.
Die Spitzen der Arme zeigen die größte Beweglichkeit.
Dieses Bild von TF1 demonstriert die Beweglichkeit der Arme. Die Daten wurden
durch Röntgenbeugung eines Proteinkristalles gewonnen. Im Kristall hat jedes Atom
eine Unschärfe, die aus relativ unterschiedlichen Lagen in den individuellen
Molekülen resultiert. Diese Unschärfe ist in der Proteindatenbank als
"Temperaturfaktor" abgelegt. Im Bild sind "kalte" Atome (= geordnet, unbeweglich)
blau dargestellt; je "wärmer" eine Gruppe ist, desto "roter" ist sie abgebildet.
Die Spitzen der Arme zeigen die größte Beweglichkeit.
Das Protein IHF (integration host factor) wurde ursprünglich als notwendig für die Integration des Phagen lambda in das Wirtschromosom gefunden. IHF wirkt außerdem an verschiedenen Stellen des Genoms je nach Kontext als positiver oder negativer Effektor auf die Transcription (Übersicht s. N Goosen, P vd Putte, Mol. Microbiol. 16 (1996) 1-7). IHF hat in allen Fällen die Funktion, die DNA in eine Form zu biegen, die anderen Proteinen ein Angreifen an spezifischen Stellen ermöglicht. IHF ist zusammen mit einem Konsensus-DNAfragment kristallisiert worden, so daß die Wechselwirkung mit der DNA hier am besten gezeigt werden kann.