Die Abbildung ist ein Screenshot der interaktiven Karte der KEGG: Kyoto Encyclopedia of Genes and Genomes. An diesem Beispiel wird demonstriert, wie die KEGG-Datenbank aufgebaut ist. In Form eines INTERFACE zu ihr wird gezeigt, wie man einen vollständigen Zugang zu den Daten dieser Datenbank - d.h. zu allen am Stoffwechsel beteiligten Enzymen sowie den Intermediär- und Endprodukten erhalten kann.
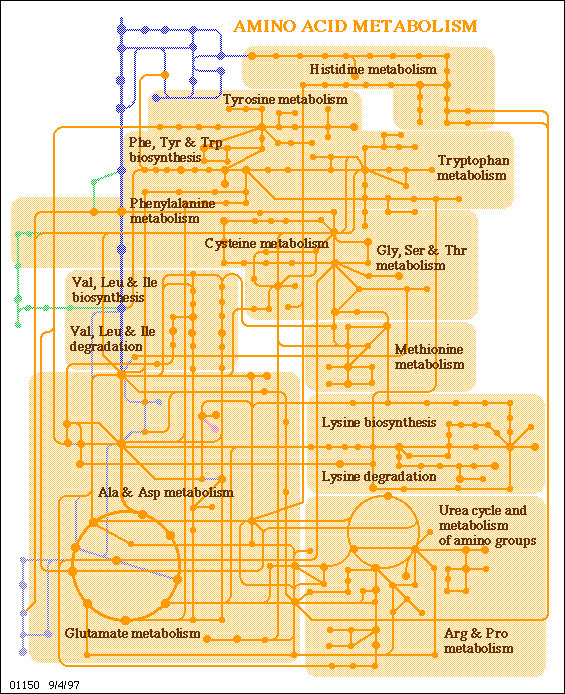
Wie arbeitet man mit der Karte? - ein Beispiel: Durch Anklicken von Lysine Biosynthesis erhält man einen Kartenausschnitt.
Nachfolgend die vollständige Liste aller bekannten Daten über Aminosäuresynthesen aus der KEGG-Datenbank (alle Links führen direkt zur Datenbank in Kyoto/Japan!)
|
|