Wie jede physikalische Struktur, können Chromosomen und damit auch Gene verändert werden. Die Änderungen machen sich als Erbänderung (Mutation) bemerkbar. Der ursprüngliche Zustand kann meist nicht wieder hergestellt werden. Wenn eine Erbinformation, etwa durch Verlust eines Chromosomenstücks, verlorengegangen ist, bleibt der Verlust irreversibel. Eine durch Mutation hervorgerufene Änderung bleibt in allen nachfolgenden Generationen erhalten, sofern der Schaden nicht Letalität zur Folge hat. An Riesenchromosomen konnte eine Anzahl verschiedenster Chromosomenänderungen (Chromosomenmutationen) identifiziert werden, die, wie sich später zeigte, auch für andere Chromosomen gelten:
Defizienz. Stückverlust an einem der Enden. Da der Stückverlust fast immer nur in einem der beiden homologen Partner erfolgt, erhält man Paarungen zwischen einem defekten und einem intakten Chromosom. Defizienzen sind im cytologischen Bild an einer Stufe erkennbar. Der nicht gepaarte Abschnitt des intakten Partners ist ein gutes Indiz für die Länge des verlorengegangenen Stückes.
Deletion. Stückverlust in der Mitte eines Chromosoms. Einen Verlust in der Chromosomenmitte erkennt man an einer Ausstülpung. Dem defekten Partner fehlt nämlich der Abschnitt, der beim intakten vorhanden ist. Aus der Länge der Ausstülpung kann wiederum auf die Lage der Deletion und ihren Umfang geschlossen werden.
Duplikation. Einzelne Chromosomenabschnitte sind verdoppelt. Auch hier kommt es bei der Paarung zu einer Ausstülpung. Um eine solche von einer Deletionsschlaufe zu unterscheiden, muß man das Bandenmuster analysieren. Liegt im nicht gepaarten Abschnitt ein Muster vor, das sich an anderer Stelle (meist benachbart) wiederholt, hat man es mit einer Duplikation zu tun. Fehlt ein Abschnitt, liegt eine Deletion vor.
Inversion: Hierbei handelt es sich um eine Umkehr eines bestimmten Chromosomenabschnitts. Die Paarung homologer Chromosomen ist unter Bildung von oft recht komplex erscheinenden Schleifen (Schlaufen) möglich.
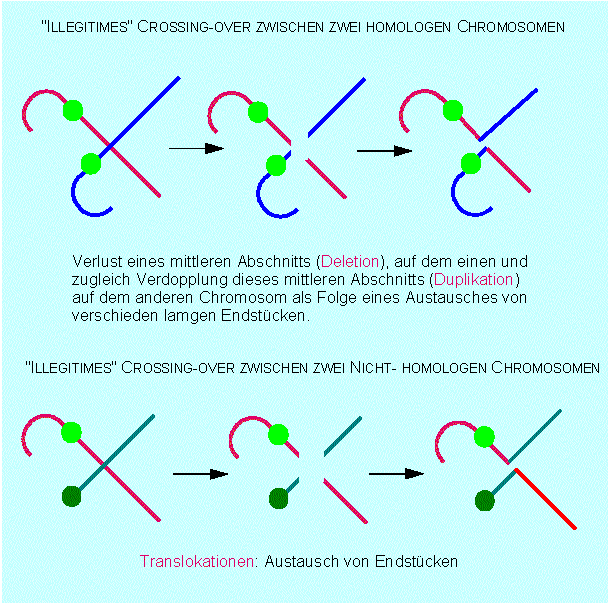
Translokationen. Unter Translokation versteht man die Übertragung eines Chromosomenabschnitts (Segments) von einem Chromosom auf ein anderes (nicht homologes). Translokationen sind bei einer Anzahl von Arten (z.B. Mais, Oenothera u.a.) nachgewiesen worden.
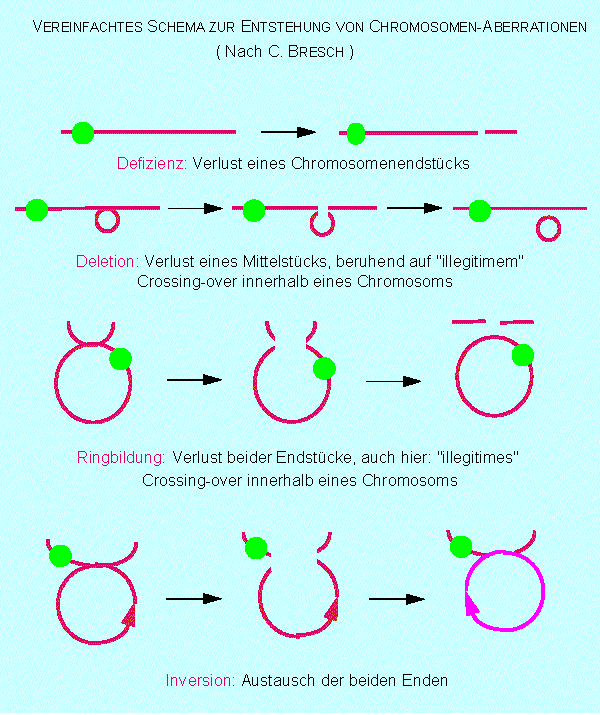
Wie entstehen Chromosomenmutationen? Am einfachsten sind die Defizienzen erklärbar. Sie entstehen als Folge eines einfachen Chromosomenbruchs. Zahlreiche mutationsauslösende Agentien, z.B. bestimmte Chemikalien oder ionisierende Strahlung, induzieren solche Schäden, genauer gesagt, sie erhöhen die Wahrscheinlichkeit des Auftretens von Brüchen. Chromosomenbruchstücke ohne Centromer gehen in der nachfolgenden Mitose verloren. Sie werden in keinen der beiden Tochterkerne integriert, verbleiben meist in der Ebene der Äquatorialplatte und gehen dort zugrunde.
Die übrigen Chromosomenmutationen sind durch zwei unmittelbar aufeinander folgende Ereignisse erklärbar: Bruch, dann Verwachsung (Fusion). Dieser Mechanismus liegt auch dem Crossing-over zugrunde. Rein formal lassen sich Deletion und Duplikation durch ein "illegitimes" Crossing-over verstehen. Beim "normalen" Crossing-over entstehen zwei Chromatiden, die den ursprünglichen homolog sind (d.h. gleich lang und mit der gleichen Zahl an Genorten). Beim "illegitimen" Crossing-over hingegen entstehen zwei ungleich lange Produkte. Als Folge davon verliert das eine Chromatid ein Stück (Deletion), das zweite gewinnt eines hinzu, und zwar einen Abschnitt, den es eigentlich selbst schon besitzt (Duplikation). Meist liegen duplizierte Abschnitte nebeneinander (als Tandem).
Das klassische Beispiel für eine solche Deletions- / Duplikations-Entstehung ist das Bar-Gen (der Bar-Locus; wegen der Dominanz über den Wildtyp wird "Bar" groß geschrieben) von Drosophila. Drosophila besitzt normalerweise annähernd oval geformte Augen. Bei der Mutante Bar sind sie, sofern das Gen in homozygotem Zustand vorliegt, balkenförmig (= Bar). Die Erscheinung Bar selbst beruht bereits auf einer Duplikation eines Chromosomenabschnitts. Die Anlage für dieses Merkmal kann einer bestimmten Chromosomenbande zugeordnet werden. Durch "illegitimes" Crossing-over im Bereich dieses Abschnitts kann aus dem Komplex einmal durch Stückverlust der Normalzustand wiederhergestellt werden (Rückmutation), zum anderen entsteht im homologen Chromatid ein Abschnitt, in dem die Chromosomenbande dreimal hintereinandergeschaltet ist (> Doppelbar).
Duplikationen sind für den Organismus keineswegs folgenlos. Die Ausprägung des Merkmals Bar zeige ja schon, daß nicht das Fehlen oder der Defekt eines bestimmten Gens die abnorme Augenform hervorruft, sondern ein überzähliger Chromosomenabschnitt.
In einem normalen diploiden Genom gibt es für jedes Gen zwei Allele. Sie liegen auf getrennten (aber homologen) Chromosomen, und man sagt daher, sie seien in der trans-Konformation. Nach Duplikation eines Chromosomenabschnitts erscheinen die darauf liegenden Gene nunmehr vierfach, je zweimal in trans und in cis (cis = auf demselben Chromosom):
A B C C D E F . . .
C und C werden nunmehr als Pseudoallele bezeichnet. Sie werden
während der meiotischen Segregation (Trennung homologer Chromosomensätze)
nicht voneinander getrennt, es sei denn durch Crossing-over. Ein Allel
und das zugehörige Pseudoallel können voneinander unabhängige
Mutationen akkumulieren und damit eine voneinander unabhängige Evolution
durchlaufen. Nur selten ist die Wirkung von Allel und Pseudoallel additiv.
Ein Pseudoallel (in cis) ersetzt damit nicht ein zweites Allel (in trans).
Diese Beobachtung weist darauf hin, daß Gene in einem Genom keine
selbständigen Einheiten sind, sondern daß ihre Aktivitäten
durch benachbarte Genabschnitte gesteuert werden. Ursprünglich nannte
man diese Erscheinung Positionseffekt. Die molekularbiologische Analyse
beginnt, Aufschlüsse darüber zu geben, wie die Kontrollmechanismen
einzelner Gene aussehen könnten.
Die Entstehung einer Inversion wurde von
B. McCLINTOCK (1938) am Beispiel der Maischromosomen analysiert. Nach dem
Zerbrechen verkleben einander homologe Abschnitte miteinander, mit der
Folge, daß neben centromerenfreien Stücken (die als Verlust
abzuschreiben sind), solche mit zwei Centromeren entstehen. Während
der Anaphase wandern diese zu entgegengesetzten Polen, so daß der
zwischen ihnen liegende Abschnitt eine Brücke bildet, die schließlich
reißt. Da der Ort des Bruches dem Zufall überlassen ist, wird
eine der Tochterzellen ein verlängertes Chromosomenstück, die
andere ein verkürztes erhalten. Im verlängerten ist der zusätzliche
Bereich zum bereits vorhandenen invertiert.
Translokationen entstehen als Folge einer Paarung nichthomologer Chromosomen während der Meiose. Oenothera ist das klassische Beispiel hierfür. Wir haben bereits gesehen, daß Oenothera lamarckiana nur als Hybrid lebensfähig ist. Im Gegensatz zu den meisten anderen Tier- und Pflanzenarten sind bei Oenothera die Kopplungsgruppen und Chromosomen nicht miteinander korrelierbar. Es gibt n = 7 Chromosomen, doch nur eine Kopplungsgruppe (= Komplex, alternativ gaudens oder velans). Die Ursache hierfür liegt in einer Serie regelmäßig aufeinanderfolgender Translokationen (R. E. CLELAND, University of Indiana, Bloomington, 1949).
CLELAND symbolisierte den Informationsgehalt eines jeden Chromosoms durch zwei von einem Punkt getrennte Ziffern. Der mütterliche Chromosomensatz wäre dann wie folgt zu schreiben :
1.2 3.4 5.6 7.8 9.10 11.12 13.14
Dem steht der väterliche Chromosomensatz gegenüber:
2.3 4.5 6.7 8.9 10.11 12.13 14.1
Während der Meiose paaren sich die homologen Chromosomenabschnitte. In der Regel sind sie ja mit einander homologen Chromosomen identisch. Hier ist es anders, denn aus den dargestellten Chromosomen bildet sich aufgrund der regelmäßigen (balancierten) Translokationen ein Chromosomenring aus . Während der Anaphase I werden die Centromeren streng alternativ auf die eine und die andere Tochterzelle verteilt. Das Ergebnis: es bleibt alles beim alten. Die Chromosomen des einst mütterlichen und des einst väterlichen Chromosomensatzes bleiben geschlossen beieinander, und das erklärt das Vorliegen von nur einer Kopplungsgruppe.
Dieses System funktioniert in der Regel zwar ganz gut, doch ist es störanfälliger als der übliche Meiosemechanismus. Die Störungen manifestieren sich in einer erhöhten Mutationsrate innerhalb der Gattung Oenothera. Das ist auffällig genug und ist wohl auch der Grund dafür gewesen, daß H. de VRIES zu Beginn des Jahrhunderts das Phänomen der Mutation gerade bei diesen Arten entdeckt hatte. Vergleichbare Situationen wurden u.a. bei Rhoeo discolor, Paeonia californica sowie bei Arten aus der Gattung Datura nachgewiesen.
|
|