1892 fand der Russe D. IWANOWSKI, daß sich die Mosaikkrankheit
von Tabakpflanzen durch einen bakterienfreien Preßsaft aus kranken
Tabakblättern auf gesunde übertragen läßt (horizontale
Transmisson). Sechs Jahre später (1898) wurde der Befund durch den
Holländer M. W. BEIJERINCK bestätigt. Er filtrierte den Extrakt
aus kranken Pflanzen durch bakteriendichte Porzellanfilter und fand, daß
dadurch keine Infektionseinbuße zu verzeichnen war. Wie schon erwähnt,
wurde das Virus 1935 isoliert und kristallisiert. 1937 wiesen F. C. BAWDEN
und N. W. PIRIE (Rothamsted Experimental Station, England) nach, daß
die Präparate Phosphat enthalten.

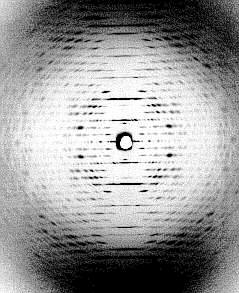
Links: Kristalline Anordnung von Partikeln des Tabakmosaikvirus in Mesophyllzellen des Tabaks. Seitenansicht (Fischgrätenmuster) (J. H. M. WILLISON, Halifax, 1976). Rechts: Diffraktionsbild (Röntgenstrukturanalyse - Faserdiagramm) einer hochkonzentrierten TMV-Lösung, in der alle Viruspartikel parallel zueinander angeordnet sind. (K. C. HOLMES, Heidelberg, 1970)
Es gibt eine Anzahl von TMV-Wildstämmen, die sich u.a. durch ihr Wirtsspektrum und die Primärstrukturen des Hüllproteins voneinander unterscheiden. Der klassische, sich am besten auf Tabak vermehrende Stamm heißt vulgare. Ein Stamm aus Tomaten, der sich aber ebenso gut auf Tabak vermehren läßt, wurde dahlemense genannt (G. MELCHERS, seinerzeit Kaiser-Wilhelm Institut für Biologie, Berlin-Dahlem). Ein dritter Stamm - aus Wegerich gewonnen - ist als Holmes Ribgrass (HRG) in die Literatur eingegangen (F. O. HOLMES, 1934).
Die einzelnen Stämme sind auf Tabakpflanzen durch ihre unterschiedlichen Symptome klar voneinander zu unterscheiden. Durch den Vergleich von Aminosäuresequenzen der Hüllproteine von vier Wildstämmen wurde deutlich, daß ein bestimmter Bereich stets unverändert ist. Es war daher zu vermuten, daß es sich um jenen Abschnitt des Proteins handelt, der in direktem Kontakt zur RNS steht. Nach Aufklärung der Tertiärstruktur bestätigte sich diese Annahme.
Wie bereits gesagt, konnte durch Behandlung mit einem Mutagen eine Anzahl von Mutanten erzeugt werden, deren Protein sich in einer oder zwei der 158 Aminosäuren vom Wildstamm unterschied. Die Existenz eines so großen Sortiments von Proteinen, die sich nur wenig voneinander unterscheiden, war der Ausgangspunkt zur Klärung einer Reihe weiterer Probleme:
Genügt die Veränderung einer von 158 Aminosäuren, um den Austausch serologisch nachzuweisen?
Sind die Austausche statistisch über die gesamte Länge der Polypeptidkette verteilt, oder gibt es bevorzugt variable Abschnitte?
Wie wird die Stabilität der Polypeptidkette nach Austausch einzelner Aminosäuren verändert?
Beeinflußt eine Veränderung des Hüllproteins die Krankheitssymptome einer Tabakpflanze?
Zu 1. Es gibt eine Kreuzreaktion aller Mutanten mit einem Antiserum, das gegen den Wildstamm gerichtet ist; der Grad der Kreuzreaktion kann jedoch von Fall zu Fall verschieden sein. Es gibt Mutanten mit Austauschen, die serologisch nicht vom Wildstamm zu unterscheiden sind. Es gibt andere, bei denen der Unterschied deutlich zutage tritt. Ein Austausch von Pro > Leu in Position 20 ist serologisch nicht nachweisbar, der gleiche Austausch in der Position 156 macht sich aber deutlich bemerkbar. Die Befunde dieser Versuchsserie ließen den Schluß zu, daß nur solche Aminosäureaustausche zu serologisch erkennbaren Unterschieden führen, die direkt an der Partikeloberfläche lokalisiert sind oder die indirekt die Konformation der Partikeloberfläche verändern. Die röntgenstrukturanalytische Aufklärung der Tertiärstruktur brachte eine Bestätigung der 1965 aufgestellten Annahme.
Zu 2. Die durch Mutagene erzeugten Veränderungen in der Polypeptidkette sind nicht zufallsgemäß über die Gesamtlänge verteilt, sondern sie konzentrieren sich in bestimmten Abschnitten. Es sieht demnach so aus, als ob Austausche in anderen Bereichen die Tertiärstruktur so stark verändern würden, daß keine stabile Quartärstruktur mehr ausgebildet werden kann, die RNS damit vor RNAsen der Pflanzen nicht hinreichend geschützt wäre, und daß solche Mutanten folglich nicht lebensfähig wären. Unter besonderen experimentellen Bedingungen sind Mutanten mit defektem und andersartig aggregierend`m Hüllprotein entdeckt worden.
.....................................10 .....................................20 SER TYR SER ILE THR THR PRO SER GLN PHE VAL PHE LEU SER SER ALA TRP ALA ASP PRO ............................................................................LEU .....................................30......................................40 ILE GLU LEU ILE ASN LEU CYS THR ASN ALA LEU GLY ASN GLN PHE GLN THR GLN GLN ALA VAL ............SER ............................SER............................ .....................................50 .....................................60 ARG THR VAL VAL GLN ARG GLN PHE SER GLN VAL TRP LYS PRO SER PRO GLN VAL THR VAL ....................GLY.................................................ILE.... .....................................70 .....................................80 ARG PHE PRO ASP SER ASP PHE LYS VAL TYR ARG TYR ASN ALA VAL LEU ASP PRO LEU VAL ........SER.... GLY GLY........................................................ .....................................90.....................................100 THR ALA LEU LEU GLY ALA PHE ASP THR ARG ASN ARG ILE ILE GLU VAL GLU ASN GLN ALA ALA.............................................................GLY.....ARG.... ....................................110 ....................................120 ASN PRO THR THR ALA GLU THR LEU ASP ALA THR ARG ARG VAL ASP ASP ALA THR VAL ALA ........................MET.................................................... ................................... 130 ....................................140 ILE ARG SER ALA ILE ASN ASN LEU ILE VAL GLU LEU ILE ARG GLY THR GLY SER TYR ASN ....................SER ........THR.........................ILE ....PHE.....SER ....................VAL........................................................ ....................................150.............................158 ARG SER SER PHE GLU SER SER SER GLY LEU VAL TRP THR SER GLY PRO ALA THR ............................PHE.............................LEU.....---
Aminosäuresequenz des Hüllproteins des Tabakmosaikvirus (vulgare) (erste Zeile). Unter einzelnen der Aminosäurereste sind andere Aminosäurereste eingetragen. Man findet derartige Austausche - jeden für sich - bei einer Anzahl analysierter Mutanten. Fett markierte Aminosäureaustausche lassen sich durch ein gegen den Wildtyp gerichtetes Antiserum nachweisen; Näheres hierzu im Text, s. a. Darstellung der Tertiärstruktur des Hüllproteins (H. G. WITTMANN, 1962, P. v. SENGBUSCH 1983) Noch mehr über die Quartär- und Tertiärstruktur erfahren Sie durch Anklicken an dieser Stelle.
Zu 3. Das Protein des Wildstamms aggregiert auch bei erhöhter Temperatur zu Viruspartikeln. Das Virus ist daher auch bei hoherer Temperatur (um 30o C) vermehrbar. Das Protein vieler Mutanten ist temperatursensitiv, d.h., bei erhöhter Temperatur wird keine regelmäßig gebaute Quartärstruktur ausgebildet, die Viren sind nur bei Normaltemperatur (bis etwa 20° C) vermehrbar. Sie sind folglich konditional letal (H. JOCKUSCH, Tübingen, 1964).
Zu 4. Hierzu lassen sich z.Zt. allenfalls einige bemerkenswerte Korrelationen anführen. So ist z.B. das Hüllprotein fast aller starken "Gelbstämme" positiver geladen als das entsprechende Wildstammprotein. Folgende Austausche führen zur Gelbstammbildung: Asp > Ala, Asp > Asn, Asn > Lys. Es bleibt abzuwarten, welcher Stellenwert diesen Austauschen in der Kausalkette Virusinfektion > Symptom tatsächlich zukommt, oder ob es nur Begleiterscheinungen sind.
Zur Einleitung einer Replikation in der Zelle muß die RNS von der Proteinhülle befreit werden. Die RNS (+ Strang) dient als Matrize zur Produktion eines komplementären - Strangs. Als Intermediärprodukt erscheint ein RNS-Doppelstrang. Der - Strang ist die Matrize zur Produktion zahlreicher neuer + Stränge (T. NILLSSON-TILLGREN; Institut für Genetik, Kopenhagen, 1970, 1974). Die Replikation spielt sich im Cytoplasma ab. Die Affinität der Replikase zum -Strang ist um Größenordnungen höher als zum + Strang, so daß viele +, aber nur wenige -Stränge gebildet werden.
Wie bereits angedeutet, werden Teile des + Strangs als mRNS genutzt; es werden dabei Proteine gebildet, die für die RNS-Replikation benötigt werden. Das Hüllprotein ist nicht dabei. Es zeigte sich, daß der - Strang nicht allein als Matrize zur + Strang-Bildung dient, sondern ebenso als Matrize zur Bildung eines kurzen Messengers für das Hüllprotein. Ein ähnliches Prinzip wurde beim Turnip-Yellow-Mosaikvirus (TYMV) gefunden; beim Tomato-Black-Ring-Virus (TBRV) und beim Cowpea-Virus (CPV) hingegen wird die Gesamtinformation des RNS-Strangs en bloc abgelesen. Das Transkriptionsprodukt wird anschließend proteolytisch in kleinere Polypeptide zerlegt.
Mittlerweile sind die Nukleotidsequenzen - zumindest partiell - bei einer Anzahl von Virusarten - ermittelt worden. Dabei zeigte es sich, daß sich Teile der RNS-Moleküle zu einer Sekundärstruktur falten können, die einer tRNS gleicht. Hinzu kommt, daß diese RNS in der Lage ist - jeweils artspezifisch - bestimmte Aminosäuren zu binden. Ob diese Erscheinung aber etwas mit dem Krankheitsbild der Pflanzen oder mit dem Replikationsprozeß der Viren zu tun hat, bleibt zu klären.
Interferenz: Es gilt die Regel, daß eine durch ein Virus infizierte Pflanzenzelle von keinem zweiten, ihm ähnlichen infiziert werden kann. Bei Doppelinfektionen erhält man auf Blättern daher ein Muster aus scharf gegeneinander abgesetzten Feldern, die entweder das eine oder das andere Virus enthalten. Daß zwei einander ähnliche Viren (Virusstämme des TMV) sich dennoch in einer Zelle gemeinsam vermehren können, zeigte I. TAKEBE durch Doppelinfektion von Protoplasten aus Mesophyllzellen des Tabaks. Durch Verwendung spezifischer Antikörper (indirekte Immunfluoreszenz) konnte er zeigen, daß beide Virusantigene in einer Zelle enthalten sind. Eine Doppelinfektion ist aber nur dann erfolgreich, wenn die Zelle gleichzeitig mit beiden Stämmen infiziert wird und wenn keiner der beiden Stämme dem anderen gegenüber Startvorteile erhält.
In vielen sphärischen Viren ist eine zentral gelegene, dicht gepackte Nukleinsäure von einem Proteinmantel umgeben. Die Hohlkugel (das Capsid) ist ein Polyeder, d.h., ein vielseitiger Körper. Die Zahl der Seitenflächen ist artspezifisch. Sie bilden morphologische Einheiten, die meist aus mehreren, oft gleichartigen Polypeptidketten zusammengesetzt sind. Die Kristallstrukturen von mehreren verschiedenen RNS-haltigen Pflanzenviren sind bekannt, u.a.:
Tomato Bushy Stunt Virus (TBSV) Southern Bean Mosaikvirus (SBMV) Satellit des Tabaknekrose Virus (STNV) Turnip Crincle Virus (TCV)
Alle haben eine Icosaederstruktur. Sie bestehen aus 60 (= 5 x 12) identischen Kopien gleichartiger Baumuster, die in identischer Weise zu einer Hohlkugel zusammengelagert sind. Strukturmodelle Sphärischer Viren findet man unter:
Ein einfaches Beispiel für diese Architektur ist das SBMV. Die Polypeptidkette des TBSV enthält 386 Aminosäurereste, die Tertiärstruktur mit hohem beta-Faltblatt-Anteil sieht etwas eigentümlich aus. Vier Domänen sind voneinander unterscheidbar. Der innere Teil (R) ist stets positiv geladen und reicht ins Hohlkugelinnere hinein, ein Verbindungsstück verknüpft ihn mit der Domäne S, die das eigentliche Strukturelement der Hülle (shell, daher S) bildet, und die vierte schließlich, ragt als Fortsatz (projection, P) aus der Hülle heraus und verleiht dem Virion ein stachelartiges Aussehen seiner Oberfläche. Trotz der im Vergleich zum SBMV komplexeren Struktur des Proteins bleibt das Grundmuster des Assembly erhalten. Auch hier besteht die Struktur aus drei Untereinheiten, und 60 dieser Elemente bilden ein Capsid. Die capsiduntereinheiten des Bean Pod Virus enthalten je zwei Polypeptidketten.
Eine Anzahl von RNS-Pflanzenviren enthält pro Virion mehr als nur ein RNS-Molekül (die Erscheinung ist auch bei Tierviren verbreitet; typisches Beispiel: Influenzavirus), oder das Genom ist auf verschiedene Viruspartikel verteilt. Ein Beispiel für Fall 1 ist das Cucumber-Mosaikvirus (CMV). Es enthält fünf RNS-Moleküle, von denen vier für die Replikation des Virus erforderlich sind, dem fünften wird eine Helferfunktion zugeschrieben. Es trägt exprimierbare Gene. In einem in vitro-System sind zwei Polypeptide identifiziert worden, die einen offensichtlichen Einfluß auf die Schwere des Krankheitsbildes der Pflanze nach der Infektion durch das Virus haben. Das Ausmaß ist wirtsabhängig. In Tabakpflanzen werden große Mengen dieser Satelliten-RNS (CARNA 5) gebildet. Die Menge an CMV ist reduziert, die Krankheitssymptome der Pflanze sind abgeschwächt. Im Gegensatz dazu führt die CARNA 5 nach Infektion von Tomaten mit CMV zu der letalen Tomaten-Nekrosis-Erkrankung. Aus Material einer Epidemie in Frankreich wurden große Mengen an CARNA 5 isoliert (J. M. KAPER, US. Department of Agriculture, Beltsville, Maryland, 1977).
Fall 2: Unvollständige Viren, Satelliten-Viren: Vielfach entstehen bei einer Virusvermehrung unvollständige Partikel. TMV-Präparate sind meist voll von Partikeln variabler Länge, die allesamt nicht infektiös sind. Vielfach enthalten sie anstelle der TMV-RNS - oder Bruchstücken von ihr - zelluläre RNS. Wegen dieses oft hohen Anteils nichtinfektiöser Partikel ist es sehr schwierig, die Infektionsrate (plating efficiency) des Virus zu bestimmen.
Das Genom des Alfalfa-Mosaikvirus (AMV, Luzerne-Mosaikvirus) besteht aus drei RNS-Molekülen, von denen jedes in einem anderen Capsid enthalten ist (B, M, Tb-Partikel). Zusammen sind die drei Partikel infektiös; die aus ihnen isolierten RNS-Moleküle allein sind es nicht, doch nach Zugabe des Hüllproteins gewinnen sie ihre Infektiosität zurück. Offensichtlich wird es zur Einleitung des Infektionsprozesses benötigt.
Bei Satelliten-Viren ist die Infektiosität eines Partikels von der Anwesenheit eines Helfervirus abhängig. Ein typisches Beispiel sind das Tabak-Nekrosisvirus (TNV) und sein Satellit (STNV). Die Vermehrung von TNV ist nicht auf die Anwesenheit von STNV angewiesen. TNV allein produziert in Tabakpflanzen große Läsionen. Bei Zugabe von STNV erscheinen nur kleine, denn offensichtlich inhibiert das STNV die Ausbreitung von TNV (B. KASSANIS, 1962).
|
|