 |

|
Friedrich MIESCHER geb. am 13. August 1844 in Basel, entdeckte 1869 die Nukleinsäuren. Die Arbeiten wurden im Laboratorium von Felix HOPPE-SEYLER durchgeführt, das seinerzeit im Tübinger Schloß untergebracht war. Er zeigte auch, daß die Atmungsaktivität von der CO2-Konzentration des Blutes abhängt. 1872 erhielt er eine Professur an der Universität Basel. Er starb am 26. 8. 1895 in Davos.
Nukleinsäuren sind Polymere aus Nukleotiden, die über Phosphodiesterbindungen miteinander verknüpft sind (Polynukleotide). Je nach Zuckertyp in den Nukleotiden (Ribose oder Desoxyribose), unterscheidet man zwischen den beiden Nukleinäureklassen
![]() Ribonukleinsäure (RNS, RNA) und
Ribonukleinsäure (RNS, RNA) und
![]() Desoxyribonukleinsäure (DNS, DNA).
Desoxyribonukleinsäure (DNS, DNA).
Die DNS ist als Träger genetischer Information bekannt, RNS hat die Funktion eines Messengers (messenger RNS) und wirkt an der Proteinbiosynthese (transfer RNS, ribosomale RNS) mit. Wie bei den Proteinen erfolgt die Verknüpfung der Monomeren zu Polynukleotiden gerichtet, allerdings handelt es sich hierbei nicht um einen Kondensationsprozeß (formal: unter Wasserabspaltung), sondern um eine Polymerisation, bei der ein Pyrophosphatrest abgespalten wird.
Zur Unterscheidung der Atome im Basen- und im Zuckeranteil, kennzeichnet man bei der Numerierung letztere mit einem (Strich). Man spricht daher von 1', 2'... (1 Strich, 2 Strich...) Die Polynukleotidkette wächst (während der Synthese) vom 5'- (5 Strich) zum 3'- (3 Strich) Ende. So geschrieben ist die Reihenfolge der Nukleotide kolinear zur Reihenfolge der Aminosäuren in einem Protein (vom N- zum C-terminalen Ende) (s. a. genetischer Code).
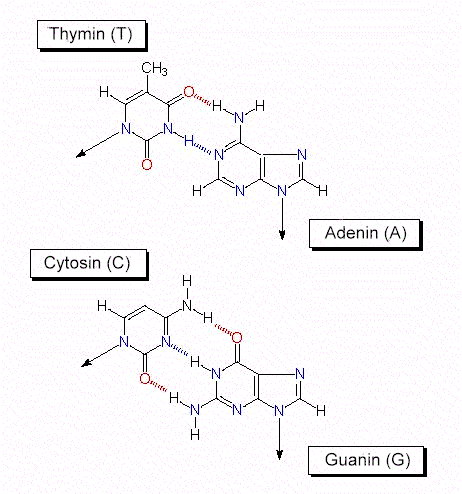
RNS und DNS unterscheiden sich außer durch ihre Zuckeranteile z.T. auch durch ihre Basen. In der RNS kommen Purin- und Pyrimidinbasen vor. In der DNS steht anstelle des Uracils ein Thymin (T). Vor allem in RNS, aber gelegentlich auch in der DNS kommen sogenannte seltene, bzw. modifizierte Basen vor. Einige von ihnen sind Intermediärprodukte der normalen Purin-, bzw. Pyrimidinsynthese.
Die Reihenfolge der Nukleotide in einem Nukleinsäuremolekül (Nukleotidsequenz) mag auf den ersten Blick willkürlich erscheinen, doch wissen wir, daß in ihr genetische Information gespeichert wird. Aber: nicht jede Nukleotidsequenz ist informationstragend. Es gibt lange, meist sich wiederholende Abschnitte in der DNS (repetitive DNS), von der wir heute noch nicht genau wissen, welche Funktionen ihnen zukommen. Seit etwa 1975 sind Methoden zur Sequenzierung von Nukleotiden bekannt.
In der DNS findet man gleich viel A wie T und gleich viel G wie C. Das Verhältnis A + T / G + C ist für jede Art spezifisch. Von diesen Befunden und von röntgenstrukturanalytischen Ergebnissen ausgehend, entwickelten J. D. WATSON und F. H. C. CRICK 1953 das nach ihnen benannte WATSON-CRICK-Modell . Sein hervorstechendstes Merkmal: Es erklärt den Mechanismus der Replikation (Reduplikation, Verdopplung) des genetischen Materials unter Ausnutzung eines jeden der beiden Stränge als Matrize. Die weiteren Eigenschaften des Modells sind:
Der Mechanismus der Replikation wurde 1958 von M. MESELSON und F. W. STAHL aufgeklärt, die eine von WATSON und CRICK gemachte Vorhersage experimentell bewiesen und damit sicherstellten, daß sich die DNS semikonservativ verdoppelt.
Im Gegensatz zur DNS ist die RNS in der Regel einsträngig. Dennoch kommen auch bei ihr doppelsträngige helicale Abschnitte vor, die darauf zurückzuführen sind, daß die Polynukleotidkette Sequenzen enthält, die sich in sich selbst zurückfalten können; sie sind spiegelsymmetrisch und bilden daher Haarnadelstrukturen (Palindrome) aus.
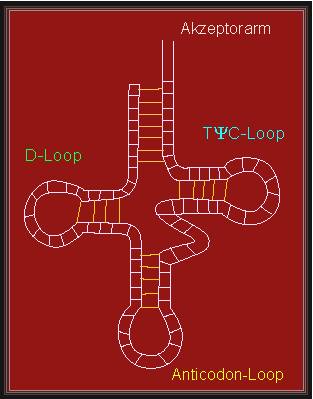
Bekannt ist die "Kleeblattkonformation" der transfer RNS (tRNS), einer Gruppe von ca. 80 Nukleotide langen Molekülen, die für die Proteinbiosynthese benötigt werden. Sie nehmen dort Adaptorfunktionen wahr. Die RNS enthält eine Anzahl "seltener" und modifizierter Basen, deren Funktion darauf beruht, zusätzliche - außergewöhnliche - Wasserstoffbrücken auszubilden und somit eine spezifische Tertiärstruktur zu stabilisieren.Details der Sekundär- und Tertiärstruktur, sowie Bedeutung der seltenen Basen:
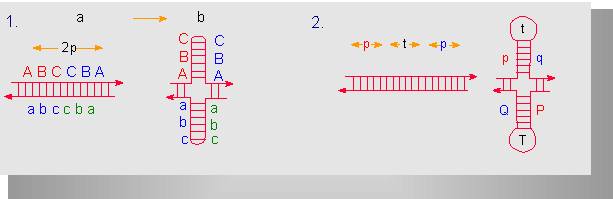
Zwei Typen invertierter (spiegelbildlicher) Repetitionseinheiten. Jede besteht aus 2p Nukleotiden, die durch Wasserstoffbrücken miteienander verknüpft sein müssen, um ein Palindrom (b) auszubilden. Bei (1) sind die Einheiten nicht durch nichtrepetitive Sequenzbereiche voneinander getrennt. Bei (2) ist das der Fall. Die Sequenz ist t Nukleotide lang. Man beachte, daß in beiden Fällen am Scheitel des Palindroms einige Basen ungepaart bleiben (Nach D. A. HAMER, C. A. THOMAS, 1974).
Komplexe
Formen der Sekundärstrukturen kommen bei der ribosomalen RNS (rRNS) vor.
Deren Aufklärung lieferte in den vergangenen Jahren stichhaltige Argumente
für Verwandtschaftsbeziehungen zwischen der ribosomalen RNS aus Chloroplasten
höherer Pflanzen und der aus Blaualgen und stellt damit eine wichtige
Stütze der Endosymbiontenhypothese dar, zum anderen belegten solche
Analysen auch die Existenz von drei Entwickungslinien zellulärer Organismen:
den Archaebakterien, den Eubakterien und jener Linie, die zur Entstehung
der Eukaryoten führte.
Weitere Abbildungen sind in der Image Library of Biological Macromolecules (IMB Jena) "with images of almost all RNA structures (hopefully) from the Protein Data Bank and Nucleic Acid Database" zu finden. Erreichbar über:
|
|