Unter Translation versteht man die Übersetzung einer Basensequenz in eine Aminosäuresequenz. Die wichtigsten Komponenten der Proteinbiosynthese haben wir bereits kennengelernt: mRNS, tRNS, Aminosäuren, ATP, GTP, Magnesiumionen, Aminoacyl-tRNS-Synthetasen und "Faktoren" (s.u.). Ferner braucht man Ribosomen.
Es gibt keine strukturelle Verwandtschaft zwischen Codon und der dazugehörigen Aminosäure, und man benötigt daher einen Adaptor (die tRNS), der die Aminosäure bindet und das zugehörige Codon erkennt. Aus der Struktur der tRNS ist folgendes ablesbar:
Das Anticodon liegt stets an der gleichen Stelle, am Ende einer der Schleifen.
Die Basen des Anticodons sind "frei", d.h. nicht mit anderen Basen innerhalb des tRNS-Moleküls gepaart. (Sie sind nicht an der Stabilisierung seiner Sekundär- und Tertiärstruktur beteiligt.)
Die Aminosäure hängt am entgegengesetzten Ende des tRNS-Moleküls an einem der freien Enden (dem 3'-Ende).
tRNS bildet klar determinierte Tertiärstrukturen aus. Die Moleküle nehmen damit eine kompakte Form an.
Die genannten Eigenschaften bilden die Voraussetzungen für
die Adaptorfunktion. Wegen der kompakten Form können an einem
mRNS-Molekül nebeneinander mehrere tRNS-Moleküle gebunden
werden, so daß Codon für Codon lückenlos besetzt
wird. Aus Stabilitätsgründen können derartige Assoziationen
in freier Lösung jedoch nicht existieren. Zur Ausbildung
einer Peptidbindung zwischen zwei Aminosäuren müssen
sie in räumliche Nähe zueinander gebracht werden. Da
ein oder mehrere Enzyme alleine dazu nicht in der Lage sind, wird
die Oberfläche einer großen supramolekularen Struktur
benötigt. Diese Aufgabe erfüllen die Ribosomen.
mRNS bindet an ein Ribosom. Aminosäurespezifische tRNS-Moleküle (Rechtecke) binden entsprechnde Aminosäuren (unter Mitwirkung spezifischer Aminoacyl-Transferasen). Der Komplex Aminosäure-tRNS wird über Codon-Anticodon-Erkennung am Ribosom gebunden. Die Aminosäure wird auf eine wachsende Polypeptidkette übertragen. Die nunmehr freie tRNS wird wieder freigesetzt.
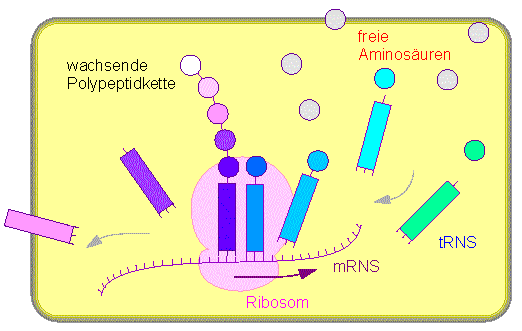
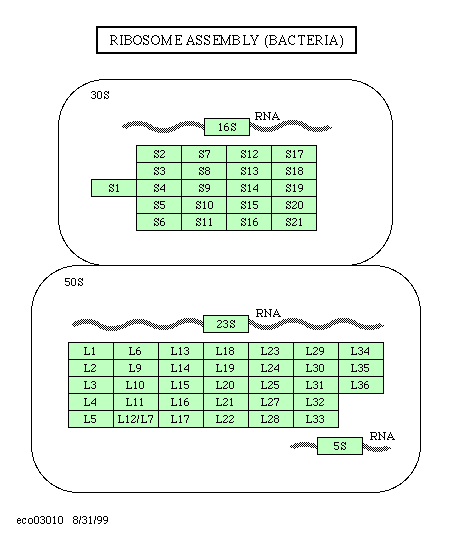
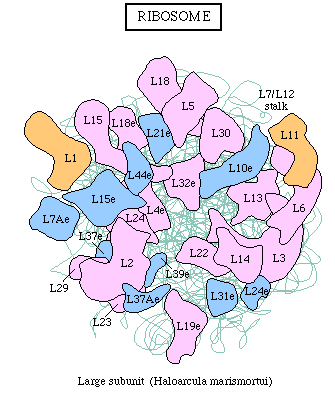
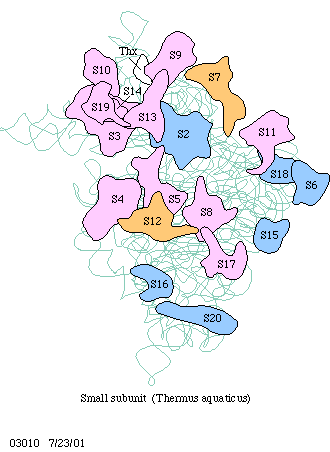
Ribosomen bestehen stets aus zwei Untereinheiten, einer großen
und einer kleinen. Jede von ihnen enthält rRNS (1-3 Moleküle)
und eine Anzahl verschiedener Proteine. Ribosomen aus Escherichia
coli sind am ausführlichsten untersucht worden. Ribosomen
aus dem Cytosol von Eukaryotenzellen sind größer als
die Prokaryotenribosomen. Zusätzlich kennt man die Ribosomen
der Chloroplasten und der Mitochondrien, die ihrerseits den Prokaryotenribosomen
ähneln.
Die Knüpfung der Polypeptidkette erfolgt selbstverständlich katalytisch. Ebenso wichtig sind die bereits genannten "Faktoren". Es sind Proteine, die für die Initiation (Start der Bildung der Polypeptidkette), die Elongation (Verlängerung der sich bildenden Polypeptidkette) und die Termination (release, dem Abbruch des Polymerisationsprozesses) benötigt werden. An jedem dieser Schritte sind ein bis drei spezifische Faktoren beteiligt. Die Faktoren aus eukaryotischen Zellen unterscheiden sich in Struktur und Funktion grundsätzlich von denen aus prokaryotischen.
Die Proteinbiosynthese ist verhältnismäßig energieaufwendig. Als energiereiche Verbindung wird neben dem ATP GTP benötigt. Ein großer Teil der Energie wird auf Kontrollmechanismen (proof-reading) verwandt. Noch vor Abschluß der Synthese beginnt sich die halb fertiggestellte Polypeptidkette zu falten, eine Sekundär- und Tertiärstruktur anzunehmen und somit in den thermodynamisch günstigsten (energieärmsten) Zustand überzugehen. Im Anschluß an die Proteinbiosynthese wird die Kette vielfach modifiziert. Es werden Teile entfernt, einige Aminosäuren verändert, Kohlenhydrate oder Lipide an bestimmte Stellen angeheftet, oder es erfolgt eine Aktivierung durch Acetylierung, Methylierung oder Phosphorylierung.
Eine Anzahl der synthetisierten Proteine verbleibt im Cytosol, andere werden durch Membranen hindurch aus der Zelle exportiert oder in Mitochondrien oder Chloroplasten importiert.
Zusammengefaßt bezeichnet man alle diese Veränderungen als Posttranslationsmodifikationen oder Modulation.
Für die Bindung einer mRNS an ein Ribosom ist eine spezifische Nukleotidsequenz, die der codierenden Sequenz vorgeschaltet ist, erforderlich. Wegen der bei Eukaryoten und Prokaryoten unterschiedlichen Erkennungssignale, "Faktoren" und weiterer Kontrollmechanismen kann eine Prokaryoten-mRNS an Eukaryotenribosomen nicht translatiert werden, ebensowenig ist die umgekehrte Version realisierbar. Daher konnte in dem zu Beginn des Themas beschriebenen NIRENBERG-MATTHAEI-System auch keine Eukaryoten-RNS exprimiert werden. Eine Expression gelang erst, als man ein entsprechendes System aus Bestandteilen von Weizenkeimlingen entwickelt hatte. Dieses Beispiel macht deutlich, wie spezifisch Translationsmechanismen sind. Vergleichbar ist die Situation bei der Transkription.
Alles (einschließlich der Posttranslationsmodifikation) zusammengefaßt, bezeichnet man als Genexpression. Die zahlreichen, dafür erforderlichen Schritte und der hohe Energieaufwand sind ein Zeichen dafür, wie wichtig diese Prozesse für den Grundstoffwechsel der Zellen sind. In Escherichia coli werden 88 Prozent des gesamten Energieumsatzes der Zelle für die Proteinbiosynthese benötigt. Der Aufwand ist zu rechtfertigen, weil die fertigen Produkte (Proteine, Enzyme, Regulatoren) Schlüsselfunktionen im Stoffwechsel der Zelle wahrnehmen, von deren Funktion der reibungslose Ablauf der einzelnen Stoffwechselwege abhängt. Dennoch ist die Fehlerquote um Größenordnungen höher als bei der Replikation. Bei der Transkription entstehen pro Gen eine Vielzahl von mRNS- und Proteinkopien. Jede Zelle kann überleben, wenn einige davon fehlerhaft sind. Bei der Replikation hingegen wird nur ein einziges Molekül DNS verdoppelt. Jede Tochterzelle erhält bei der nachfolgenden Teilung eine Kopie davon. Jeder Fehler, der dabei unterläuft, ist irreversibel und tritt als Informationsveränderung (Mutation) in allen Nachkommen jener Zellinie in Erscheinung, die eine fehlerhafte Kopie erworben hat.
Die vielen Schritte der Genexpression lassen Kontrollen auf verschiedenen Ebenen zu. Das Phänomen der Differenzierung ist ein Ausdruck differenzieller Genexpression. Es sind also die auf der DNS und von ihr auf die (m)RNS übertragenen Signale (Nukleotidsequenzen, Sekundärstrukturen), die bestimmen, ob ein Gen stärker als ein anderes exprimiert wird und daß im Verlauf der Entwicklung erst ein Satz von Genen zum Zuge kommt und später ein anderer. Umschaltungen können abrupt erfolgen, sie können aber auch fließend sein.
So hängt beispielsweise die Bindung einer mRNS an ein Ribosom von seiner Erkennungssequenz ab. Doch nicht alle Arten von mRNS enthalten die gleiche Sequenz. Folglich werden diejenigen bevorzugt an Ribosomen gebunden, deren Erkennungssequenzen die höchste Affinität zu ihnen besitzen.
Bei geringer Affinität ist die Wahrscheinlichkeit, daß mRNS an Ribosomen gebunden wird, niedrig, und als Folge davon werden nur wenige Proteinmoleküle gebildet. Aber wohlgemerkt, das ist nur eine von vielen Möglichkeiten, die Zahl der Proteinmoleküle pro Zelle zu determinieren. Eine weitere Kontrollebene ist z.B. die Affinität der mRNS zu den Initiationsfaktoren, und eine dritte wäre die Lebensdauer (Halbwertszeit) der betreffenden mRNS.
Wie die Transkription, so läuft auch die Translation nach einem Fließbandprinzip ab. Sobald ein Teil der mRNS von einem Ribosom freigegeben worden ist, wird er von einem weiteren Ribosom gebunden, so daß die Synthese einer zweiten Polypeptidkettenkopie bereits initiiert werden kann, lange bevor die erste fertiggestellt ist. Das läßt sich fortsetzen, so daß simultan an einer mRNS eine Anzahl von Polypeptidketten gebildet werden können. Es wirken dabei zahlreiche Ribosomen mit. Man nennt derartige Komplexe Polysomen.
|